農畜産物ゲノム情報データベース
イネ等作物ゲノムデータベース
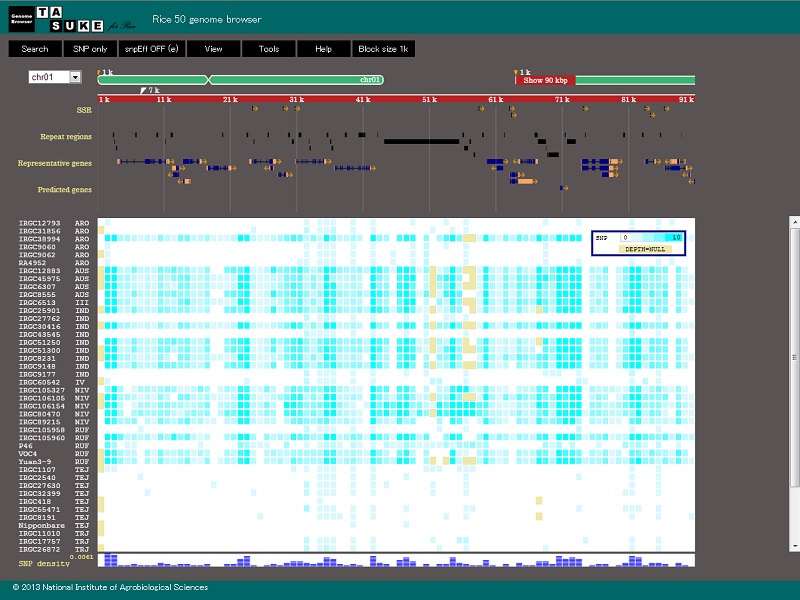 | Rice 50 genome browser |
| 生物種: Oryza sativa | |
| 説明: これはゲノムブラウザ「TASUKE」です。このブラウザは、遺伝子、繰り返し、マーカーや残りのアノテーション情報と同様に、ゲノムワイドバリアントや再シークエンシングデータのカバレッジの深さを示しています。バリアントの数とカバレッジの深さはサイズ1bpから100kbまでの可変ブロックのグラデーションカラーで示されています。 |
 | MEGANTE |
| 生物種: | |
| 説明: 植物のゲノム配列に対して遺伝子構造・機能を予測するウェブサービスです。簡単な操作で利用可能です。予測結果はゲノムブラウザ上に表示されます。Excelファイルとしてダウンロードすることも可能です。 |
 | Komugi GSP |
| 生物種: Triticum aestivum | |
| 説明: このサイトでは、生物研を中心に日本で進められているコムギゲノム解読に関する最新情報(発表論文、学会情報等)を得ることができます。また、コムギに関するゲノム配列や、遺伝子発現情報、アノテーション情報にアクセスすることができます。 |
 | bex-db |
| 生物種: barley (Hordeum vulgare) | |
| 説明: オオムギ完全長cDNA及びマイクロアレイによる発現情報に関するデータベース |
 | DAIZUbase |
| 生物種: Glycine max | |
| 説明: DAIZUbaseは、統合された大豆のゲノム·データベースとデータマイニングツールで、2マップブラウザ、遺伝子ビューア、およびBLAST検索システムから構成されています。 |
 | RiceXPro |
| 生物種: Oryza sativa | |
| 説明: Rice Expression Profile Database(RiceXPro)は天然のフィールド条件や、さまざまな植物ホルモンで処理されたイネの苗及びレーザーマイクロダイセクション(LMD)によって分離された特定のセル・タイプ/組織の元で、イネの全体の成長を包含する組織/器官のマイクロアレイ解析から得られた遺伝子発現プロファイルのリポジトリです。 |
 | RiceFREND |
| 生物種: Oryza sativa | |
| 説明: RiceFRENDは、マイクロアレイの天然のフィールド条件下での成長と発展の異なる段階で様々な組織/器官から導出されたデータ、および様々な植物ホルモンで処理されたイネの大規模なコレクションに基づいたイネの遺伝子共発現データベースです。共発現解析に使用された遺伝子発現データは、RiceXPro(マイクロアレイ分析によって得られた伝子発現プロファイルのリポジトリ)の中でアクセスすることができます。 RiceFRENDは、様々な生物学的経路かつ/または代謝過程で機能的に関連する遺伝子の同定のためのプラットフォームを提供することを目的としています。 |
 | SALAD Database |
| 生物種: Oryza sativa | |
| 説明: SALADデータベースでは、植物間のアミノ酸配列の類似性をもとにデンドログラムを作成し、機能的に似たタンパク質をクラスタリングすることができます。特徴として、保存領域(モチーフ)の有無がグラフィカルに表示されること、植物種間でのアノテーション比較が容易であること、アノテーションの類似性を指標にクラスタリングできること、保存モチーフごとに簡単に分子進化系統樹を作成できること、冗長性のない全ゲノム情報を解析していること、クラスタリングにアミノ酸置換マトリックスを用いていることなどが挙げられます。最新版では、生物種10種間で比較ができます。 |
 | FiT-DB |
| 生物種: Oryza sativa | |
| 説明: FiT-DB(Field transcriptome database)は、圃場におけるイネ(日本晴-農林8号)の葉の遺伝子発現データと、その変動の背景にある要因の統計モデリング解析結果のデータベースです。それぞれの遺伝子の発現が、気象条件(気温・日射量など)、概日時計、日齢からどのような影響を受けているかを調べることが出来ます。また、逆に「気温の影響を受けている遺伝子」のような、様々な条件から遺伝子を検索することも可能です。 本データベースでは、"What's FIT-DB"として日本語の説明も用意されています。 |
 | RAP-DB |
| 生物種: Oryza sativa | |
| 説明: イネアノテーションプロジェクト(RAP)によるイネゲノムの高精度注釈(アノテーション)情報を収録したデータベースです。2種類のアノテーションビューア(GBrowse、G-integra)があり、アノテーション情報をグラフィカルに参照できます。また、ユーザー自身の配列とイネゲノム配列の類似性検索(BLAST,BLAT)を行うことができます。 |
 | Mutant Panel |
| 生物種: Oryza sativa Japonica Group | |
| 説明: 内在性レトロトランスポゾン(Tos17)によるイネ遺伝子破壊系統の表現型と破壊された遺伝子の情報を格納したデータベースです。破壊系統に関する表現型や遺伝子の情報だけでなく、イネゲノム塩基配列上のTos17挿入位置や遺伝子の位置情報をグラフィカルに参照できます。またお手持ちの配列とTos17隣接配列の類似性検索や、破壊系統種子の配布依頼もできます。 |
 | Q-TARO |
| 生物種: Oryza sativa | |
| 説明: 文献から集めたイネのQTL情報のデータベースです。 種々の文献検索サイトよりイネのQTL研究に関した文献情報を抽出し、最終的には1214件の論文から5096件のイネQTL情報を得ました。その上で、統計上検出された遺伝子座間相互作用あるいは遺伝子×環境間相互作用のQTLは排除しました。さらに染色体上の同一カ所に検出されたQTLを整理し、QTL物理位置の同じ形質名を持ち同じインターバルに検出されたQTLを統一することによって、冗長性の少ない代表的QTL情報を得ました。代表的QTLは、463件の論文に由来する1051件のQTL情報から構成されています(2008年3月31日現在)。これらの代表的QTL情報はSQL言語を用いて構造化され、表およびゲノムブラウザから関連情報を得ることができるQ-TARO (QTL Annotation Rice Online)データベースとして整理されました。2012年現在、新たなQTL情報の追加は行っていません。 |
 | OGRO |
| 生物種: Oryza sativa | |
| 説明: 文献データベースWeb of Scienceに対して、遺伝子機能解析に用いられる用語をキーワードとして検索し抽出された文献のうち、遺伝子機能が表現型と関連づけられている文献から702の遺伝子座をイネにおける機能既知遺伝子としました(2012年3月31日現在)。最終的には、それらの情報を表現型、機能解析の方法およびゲノム位置の情報とともに、QTLデータベースであるQ-TARO (http://qtaro.abr.affrc.go.jp/) と同一のプラットホーム(表とゲノムブラウザ)で表示可能なシステムを構築し、機能既知遺伝子のゲノム上の分布とQ-TAROに登録されているQTL領域などと比較できるようにしています。2012年現在、機能遺伝子情報は更新しています。 |
 | ineweb |
| 生物種: Oryza sativa Japonica Group | |
| 説明: イネの品種情報と特性を収録したデータベースです。各品種につき、育成地、系統、交配、登録番号や年、作付面積と全国の順位、普及見込み地帯、来歴、形態的特性、生態的特性、配布種子量、栽培適地、栽培上の注意等が詳細に掲載されています。 検索方法には、「品種名検索」、「特性情報検索」、「作付面積検索」、「登録特性検索」の4種類が用意されています。 |
 | PLACE |
| 生物種: Embryophyta | |
| 説明: 高等植物遺伝子のプロモーター等の発現制御に関わる領域の塩基配列モチーフを論文から収集し、解説や文献、PubMedへのリンク、国際塩基配列データベースのアクセッション番号などを付加したデータベース。 |
 | SGD |
| 生物種: Cryptomeria japonica, Chamaecyparis obtusa | |
| 説明: 本データベースは、スギ(Cryptomeria japonica )とヒノキ(Chamaecyparis obtusa )の遺伝子の部分塩基配列(EST)、DNAマーカー、遺伝連鎖地図、遺伝的多様性などに関する情報を提供しています。 |
 | GFSelector |
| 生物種: Oryza sativa | |
| 説明: ゲノム配列から複数の解析ツールを用いて遺伝子予測を行うプログラムです。 RiceGAASで使用されているプログラムで、 概要説明とイネゲノム配列での解析結果へのリンクが掲載されています |
 | RGP |
| 生物種: Oryza sativa | |
| 説明: イネゲノムの高密度分子遺伝地図、酵母人工染色体(YAC)の整列化地図、65000個のEST塩基配列情報、及び、これらESTをYAC整列化地図上に位置づけた発現遺伝子地図が公開されています。 |
 | RGP: RICE GENETIC MAP IN NATURE GENETICS |
| 生物種: Oryza sativa | |
| 説明: イネ連鎖地図作成に使用された1,383個のRFLPマーカ情報(1993年版)が掲載されています。 |
 | RGP gmap98 |
| 生物種: Oryza sativa | |
| 説明: イネ連鎖地図作成に使用された2,275個のRFLPマーカ情報(1998年版)が掲載されています。 |
 | RGP gmap2000 |
| 生物種: Oryza sativa | |
| 説明: イネ連鎖地図作成に使用された3,267個のRFLPマーカ情報(2000年版)が掲載されています。 |
 | RGP caps |
| 生物種: Oryza sativa | |
| 説明: イネ連鎖地図作成に使用された332個のSTS/CAPSマーカ情報が掲載されています。 |
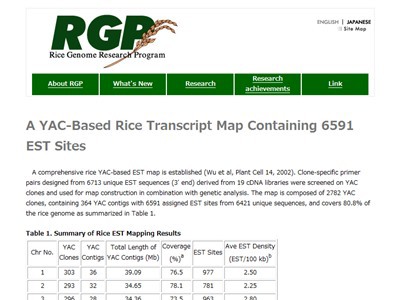 | RGP estmap2001 |
| 生物種: Oryza sativa | |
| 説明: 6,591個の発現遺伝子配列断片(EST)を酵母人工染色体(YAC)整列化地図上に位置付けたイネ発現遺伝子地図が掲載されています。 |
 | RGP physicalmap |
| 生物種: Oryza sativa | |
| 説明: 酵母人工染色体(YAC)によるイネ物理地図情報(1994~1997年版)が掲載されています。 |
 | RGP physicalmap2001 |
| 生物種: Oryza sativa | |
| 説明: 酵母人工染色体(YAC)によるイネ物理地図情報(2001年版)が掲載されています。 |
 | RGP kasalath |
| 生物種: Oryza sativa | |
| 説明: インディカ米(カサラス)のBAC情報が掲載されています。 カサラスのBACend配列やBAC物理地図情報を参照できます。 |
 | RGP chr1 |
| 生物種: Oryza sativa | |
| 説明: イネ第1染色体の概要説明と関連データへのリンクが掲載されています。 |